Molekulare Paläontologie - "Auferweckung" urzeitlicher Proteine
In-silico-Verfahren gewährt Einblick in die Eigenschaften von Proteinen aus ausgestorbenen Organismen
DOI:
https://doi.org/10.5283/bidw.v29i41.152Schlagworte:
Biologie und vorklinische Medizin, Biophysik und physikalische Biochemie, Phylogenie, In-silico-Verfahren, Molekulare Paläontologie, Proteinevolution, Aminosäuresequenz, ASR, EnzymkomplexeAbstract
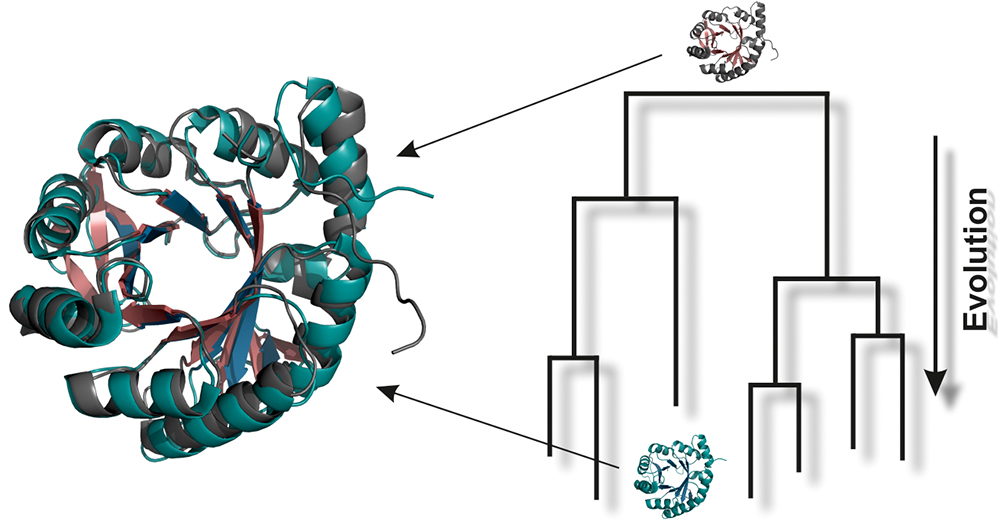
Proteine (Eiweiße) sind die wesentlichen Funktionsträger allen Lebens. Sie bestehen aus einer spezifischen, linearen Abfolge (Sequenz) von unterschiedlichen Aminosäuren. Diese Aminosäuresequenz legt eine räumliche Struktur fest, welche wiederum die Funktion eines Proteins definiert. Es ist eine interessante Frage, wie sich Aminosäuresequenzen und damit die Eigenschaften von einzelnen Proteinen im Laufe der biologischen Evolution verändert haben. In diesem Zusammenhang sind Informationen über Proteine aus dem letzten gemeinsamen Vorfahren aller heute existierenden Lebewesen (englisch: last universal common ancestor, LUCA) besonders aufschlussreich. Da der LUCA die Erde bereits vor etwa 3,5 bis 3,8 Milliarden Jahren bevölkerte, sind keine molekularen Fossilien erhalten, die es ermöglichen würden, etwas über die Eigenschaften dieser urzeitlichen Proteine in Erfahrung zu bringen. In den letzten Jahren wurden jedoch Computer-basierte Insilico-Verfahren entwickelt, welche die »Auferweckung« und Charakterisierung dieser LUCA-Proteine ermöglichen. Ihr Vergleich mit »modernen« Proteinen erlaubt Einblicke in die Gesetzmäßigkeiten der Proteinevolution und zusätzlich in den Zusammenhang zwischen der Aminosäuresequenz von Proteinen und ihrer Struktur und Funktion.
Downloads